アミノ酸配列をコードしている領域の場合は,
DNA配列をアミノ酸配列に変換することで,
正しく配列が得られているか確認できます.
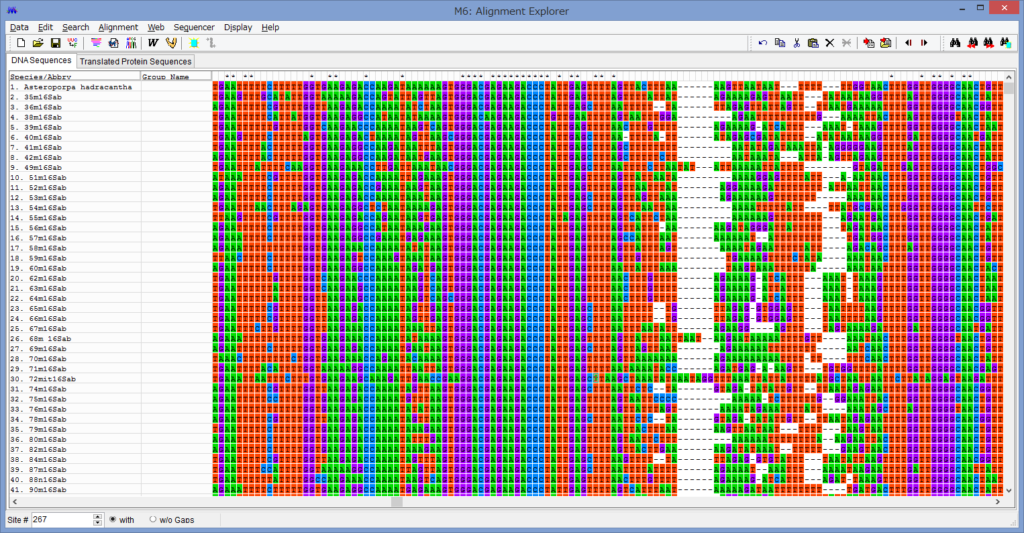
こちらは,アミノ酸をコードしているミトコンドリアのCOI領域
のDNA配列をアライメントしたものです.
ちなみに,このOTUの名前の付け方は長すぎる上に,
()とか系統解析状優しくない記号が使われている悪い例です.
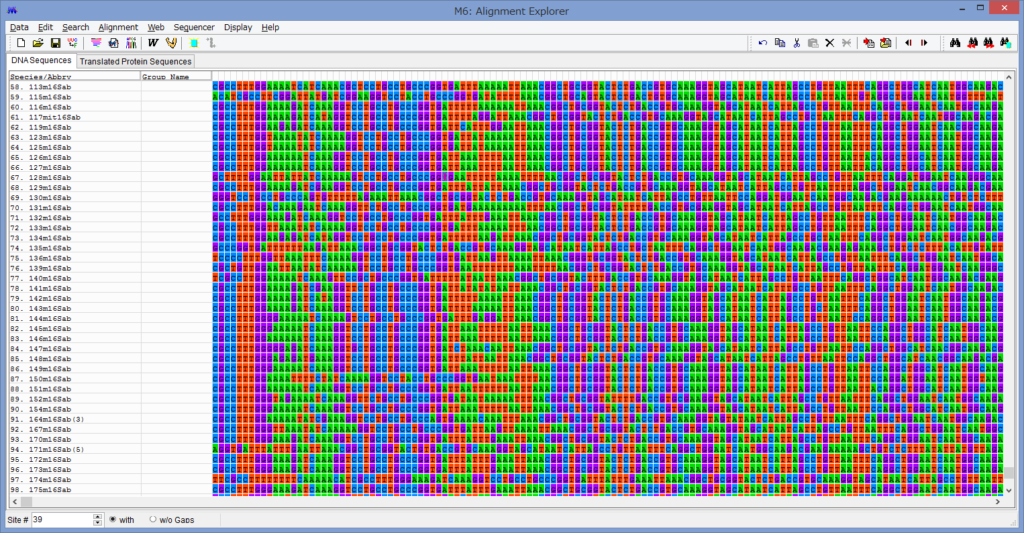
各配列の一番上の行にアスタリスク(*)が示してある列は,
全てのOTUで塩基置換が同じ事を示しています.
従って,*が無い列はどこかのOTUで塩基置換が起きています.
この*を見ると,二列おきに無くなっている傾向が見られると思います.
これは,この配列がアミノ酸をコードするコドンは塩基3つで一組となっており,
その三番目が置換しやすい事を表しています.
左上の方のDNA Sequencesの横のTranslated Protein Sequencesのタブをクリックすると,
このようなウィンドウがポップアップするので,
Yesを選択するとDNA配列がアミノ酸配列に変換されます.
Noを選択すると,
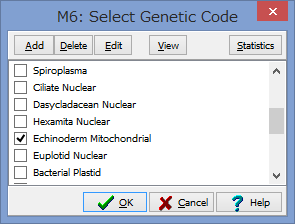
このように,コドンがコードするアミノ酸の遺伝コードを選択する事ができます.
分類群に合わせて変更しましょう.
遺伝子コード表は,Data→Select Genetic Code Tableからも選択できます.
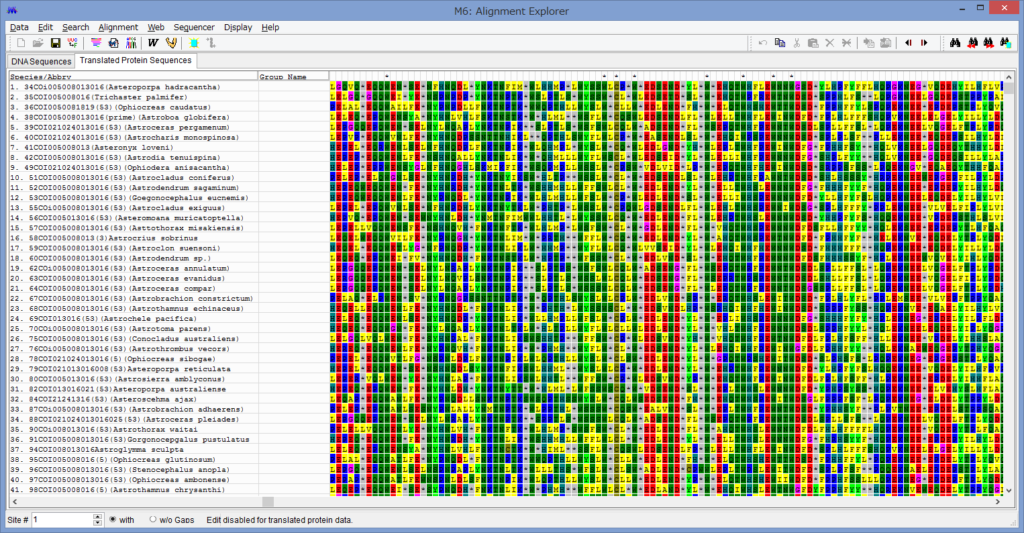
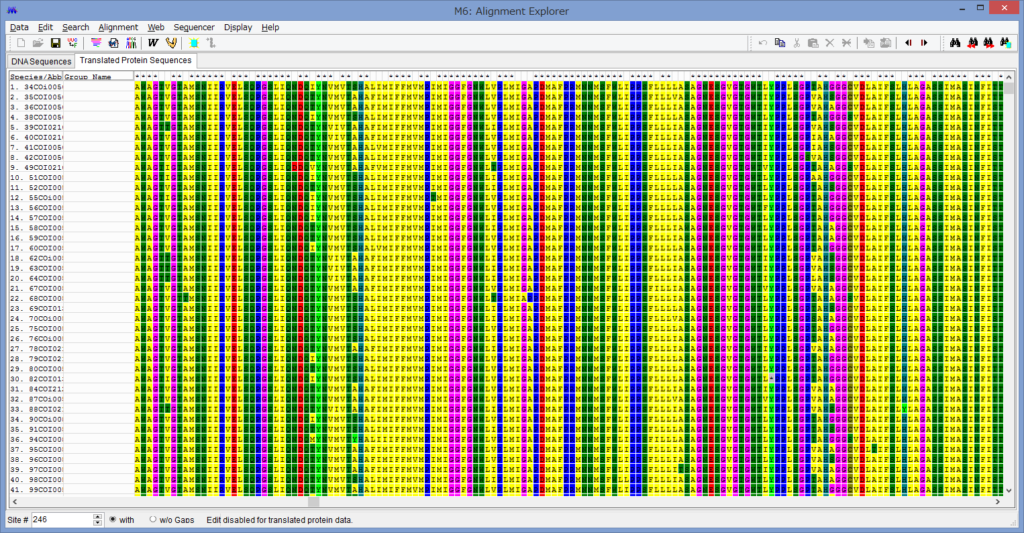
これが置換後のアミノ酸配列です.色付きの文字は全てアミノ酸です.
灰色の*は,ストップコドンでこれが見られる場合は,
アミノ酸変換が上手くいっていない事がほとんどです
(勿論,複数の遺伝子が発現するためのストップコドンの場合もあります).
というわけで,一番左の列を削除してもう一度アミノ酸変換してみました.
うーん,まだストップコドンがみられます.
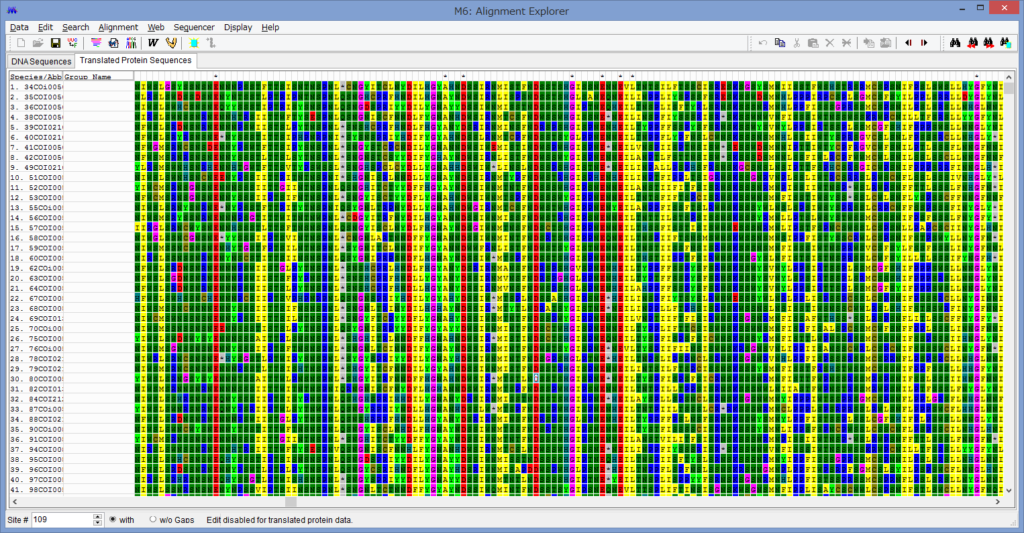
更にもう一列削除してアミノ酸変換しました.
ストップコドンもみられず,配列も結構「揃って」います.
恐らく,これで正しくアミノ酸配列に変換できたものと思われます.
今日はここまで.