※もっと良いやり方をご存知の方はそっと教えてください.
参考
http://hanzawalab.blog.fc2.com/blog-entry-43.html
http://nonomasu.blog.fc2.com/blog-entry-7.html
| 塩基配列データ作成(MEGA, DNAsP) |
MEGAでの作業
- MEGAでアライメントを行ったセッションをnexusファイルで出力する(Amo.nexusとしましょう).この段階で,AMOVA解析で扱う集団ごとにまとまるように配列の順番を入れ替えておくと後が楽.
DnaSPでの作業
- 解析用のフォルダ(Amoとしましょう)を作り,その中にAmo.nexusを入れておく.
- DnaSPを立ち上げ,File→Open Data FileでAmo.nexusを読み込む.
- Data Informationが表示されれば,読み込み成功.Data InformationをCloseで閉じる.
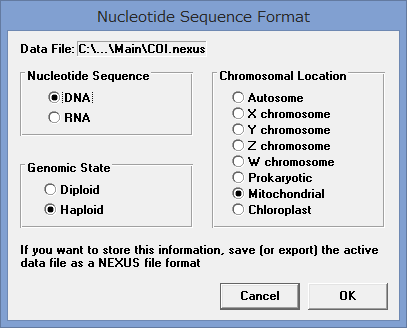
- Data→Formatでデータの形式を指定する.例えばミトコンドリアの16Sの場合は,DNA, Haploid, Mitochondorialにチェックを入れてOKをクリック.
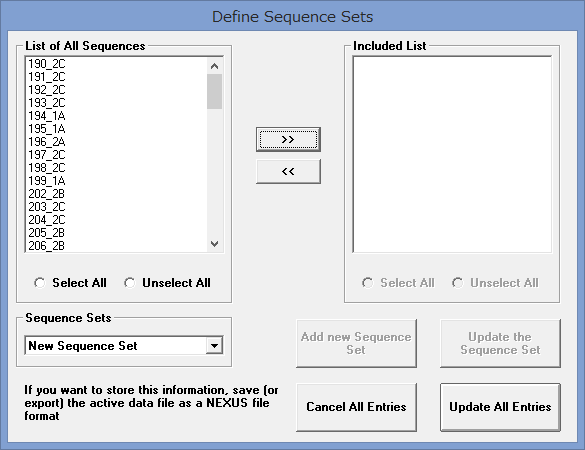
- Data→Define Sequence SetsでOTUのグループ分けを指定する.List of All Sequenesから,”>>”でIncluded ListにOTUを移動し,Add new Sequence Setで名前を付ける.
- 全てのグループに名前を付けたら,Update All Entries(赤文字になっているはず)をクリック.
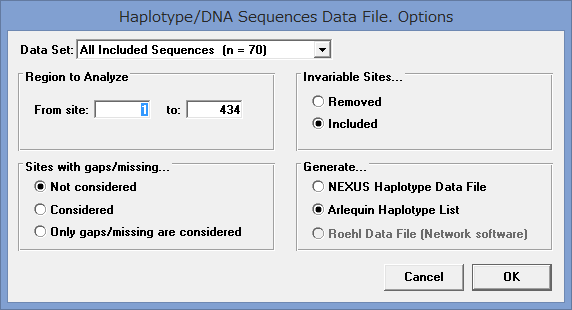
- Save/Export Data as…→Arlequin File Formatを選ぶ.
- Not considered, Included, Arlequin Haplotype Listにチェックが入っている事を確認し,OK.
- .hapと.arpの二つを保存する.Amo.hap, Amo.arpとしましょう..hapは.arpのバッチファイル的なものらしく,必ずペアでAmoに保存しておく.
- Amova解析の際には各集団を更に上位の集団にまとめる必要があるので,以下のテキストをAmo.arpの末尾に貼り付ける.赤字は任意に変えられる部分.
[[Structure]]
StructureName=”任意の名前”
NbGroups=4
Group={
“DnaSPで定義したグループ名1”
}
Group={
“DnaSPで定義したグループ名2”
“DnaSPで定義したグループ名3”
}
Group={
“DnaSPで定義したグループ名4”
Group={
“DnaSPで定義したグループ名5”
“DnaSPで定義したグループ名6”
}
- これでArlequin用の準備完了 .
※Arelquin上でもっと簡単にグループ設定する方法がありました↓
| AMOVA解析(Arlequin) |
- Arelquinを起動し,”Open project”でAmo.arpを選択.左側の枠にSamplesやGroupsが表示されたらOK.
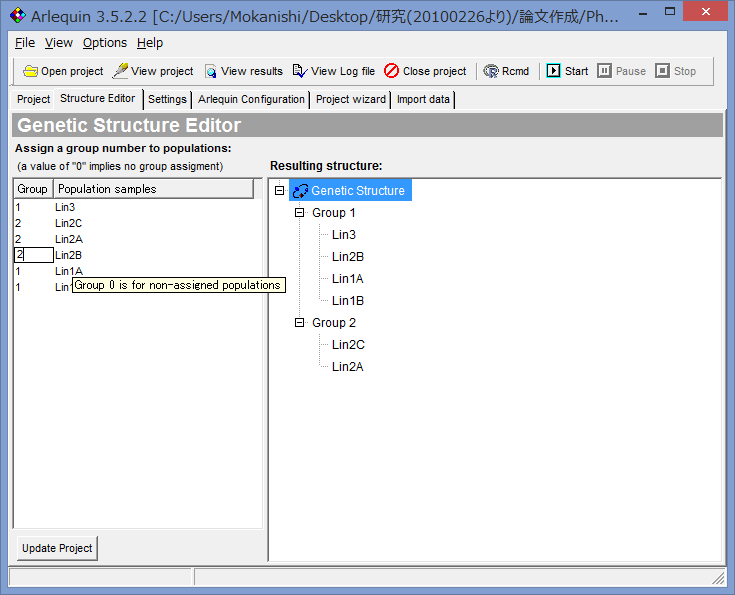
- グループを設定する場合は,Structure Editorをクリックし,Population samplesの横のGroup(デフォルトでは0が表示されている)をダブルクリックし,任意のグループ名を入れると.右側の枠に階層構造として出力される.最後はUpdate ProjectをクリックすればOK.Projectタブでグループ分けが出来たかどうか確認できる.
- “Settings”でAMOVAを選び,Standard AMOVA…をチェックし,プルダウンメニューの下の方で塩基置換モデルを選べる.普通はKimura 2Pが無難.
- “Start”をクリックして解析.うまくいけば,Amo内にログなどが入ったAmo.resというフォルダが作成される.この中のAmo.xmlにAMOVA解析結果がHTML形式で保存されている.
更新:2017/3/11