※もっと良いやり方をご存知の方はそっと教えてください.
細かいコマンドは勉強中です.こちらもいろいろ教えてください.
| 塩基配列データ作成(MEGA, BEAUti) |
MEGAでの作業
- 解析用フォルダを作る(Skyとしましょう).
- 解析したい内群について,MEGAでアライメントを行ったセッションをfasファイルで出力する(Sky.fasとしましょう).
| 各種パラメータ設定(BEAUti) |
BEAUtiでの作業
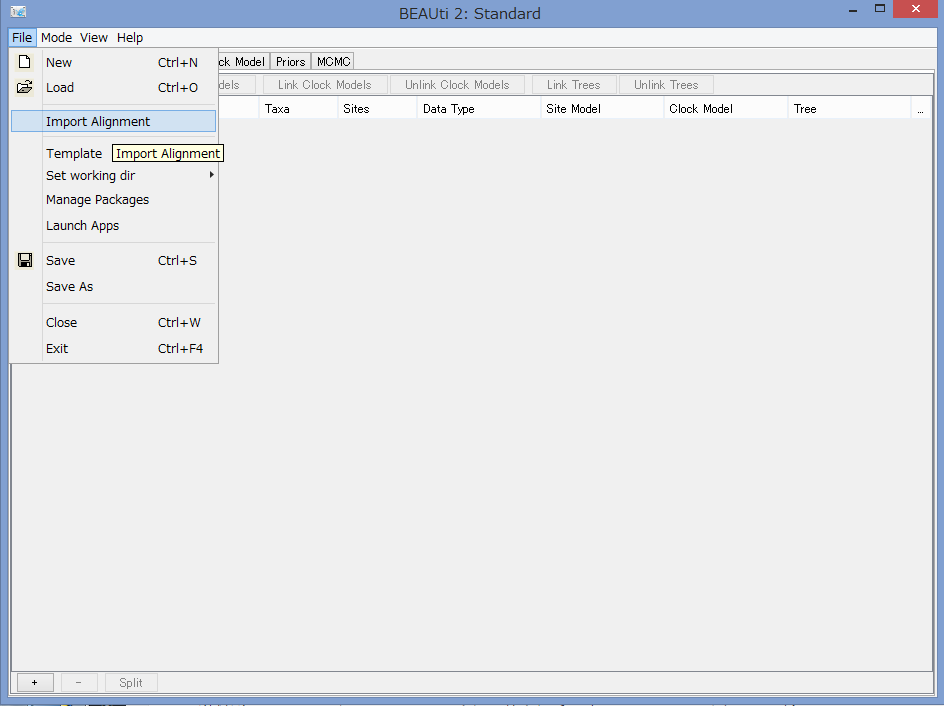
- File→Import AlinmentでSky.fasを読み込む.Partitionタブでアライメントセッションファイルの名前が確認できればOK.
- Site ModelタブのSubst Modelでモデルを設定する.
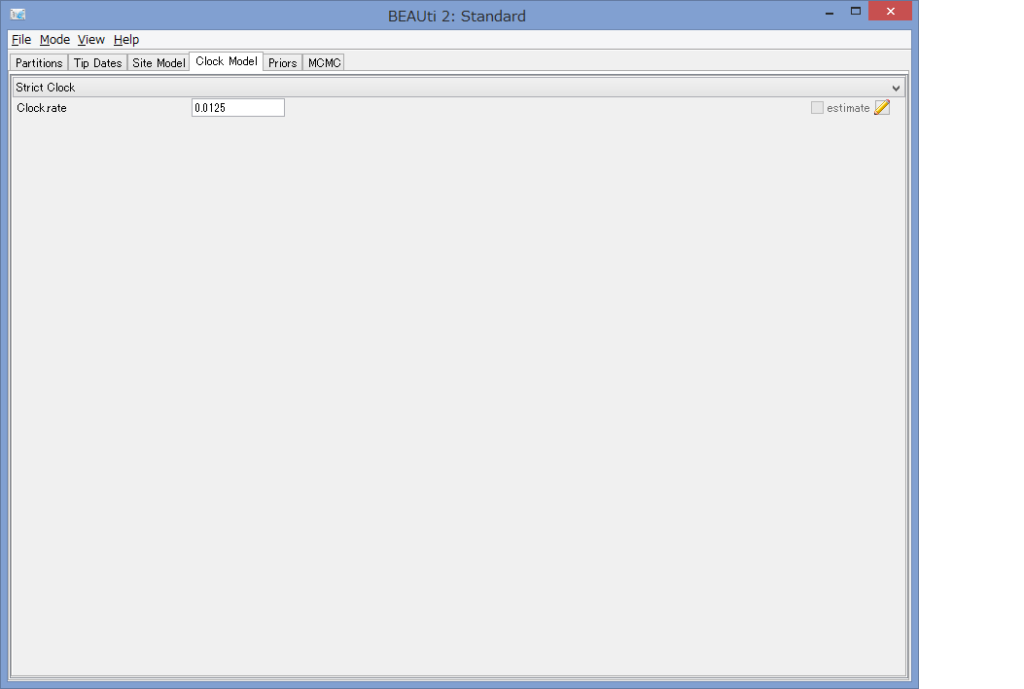
- Clock Timeタブで,特にこだわりが無ければ”Strict Clock”を選び,Clock rateに塩基置換レートを入力する.例えば,1.25%/100万年であれば,0.0125と入力.
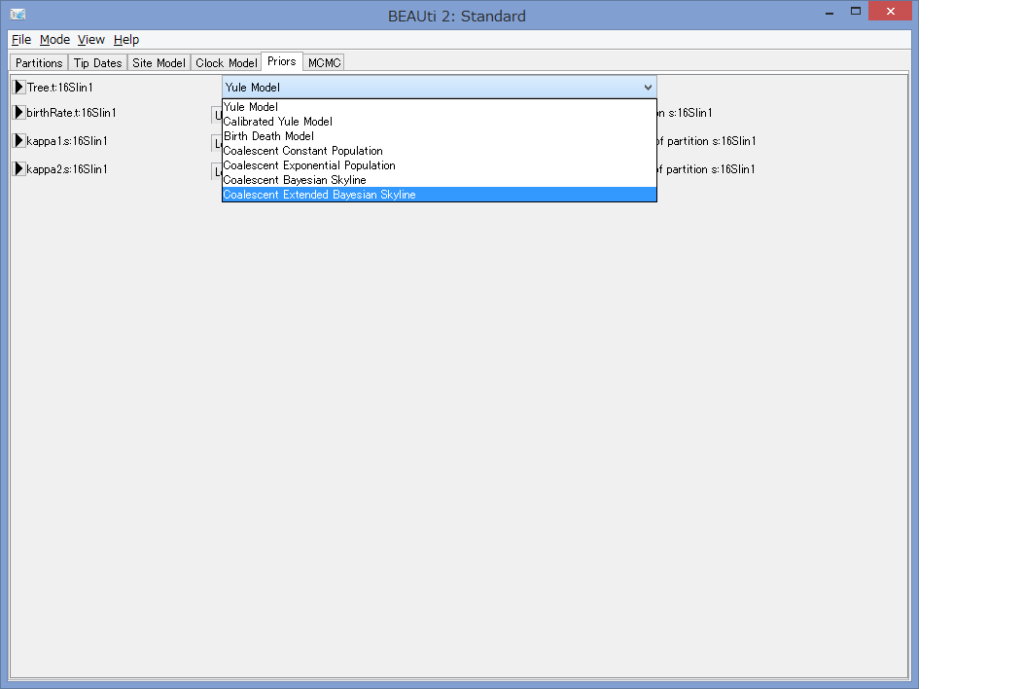
- PriorsタブでTree.t: EBSP(アライメントのセッション名)から”Coalescent Extended Bayesian Skyline”を選ぶ.
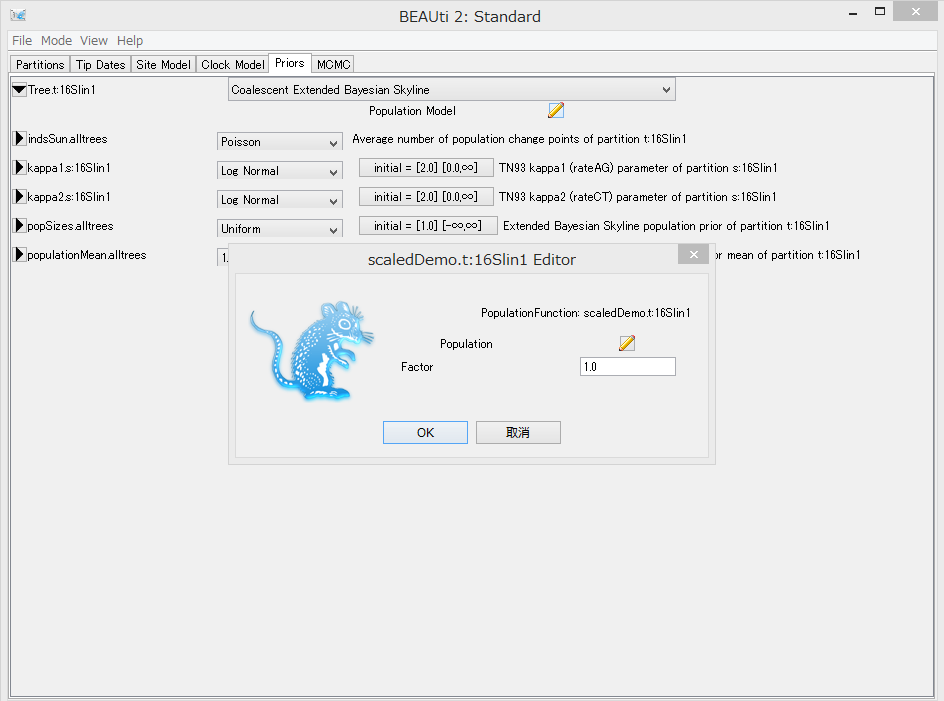
- Tree.t: EBSPの左の▶をクリックし,Population modelの横の鉛筆をクリックし,Factorを入力する.母系遺伝のミトコンドリアは.0.5と入力するのが良いようです.
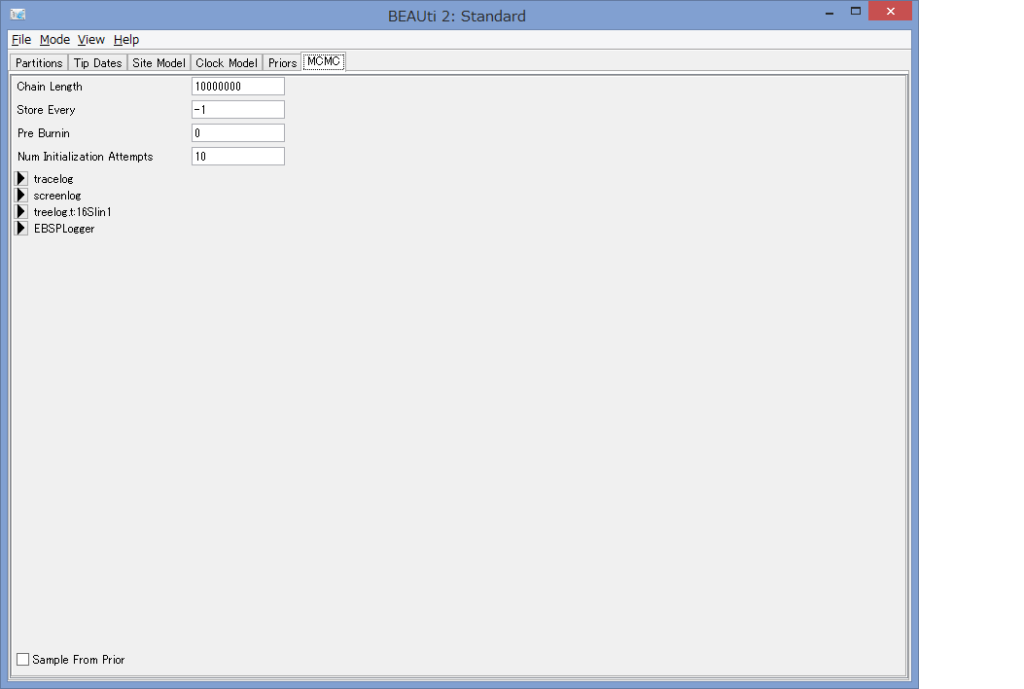
- MCMCタブのChain lengthで,MCMC連鎖の数を設定する.デフォルトは10,000,000ですが,多分30,000,000位はやったほうがよさそうです.この後のTracerで結果を見ながら,この値を調整するとよいでしょう.
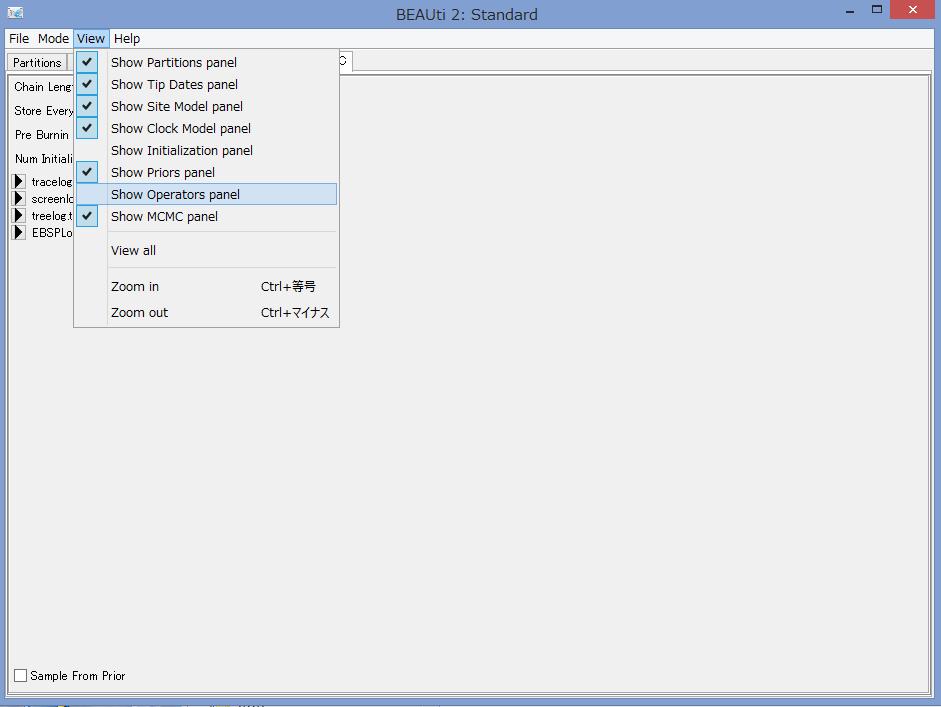
- Fileの並びのメニューのVeiwタブから,Show Operators panelを選ぶ.
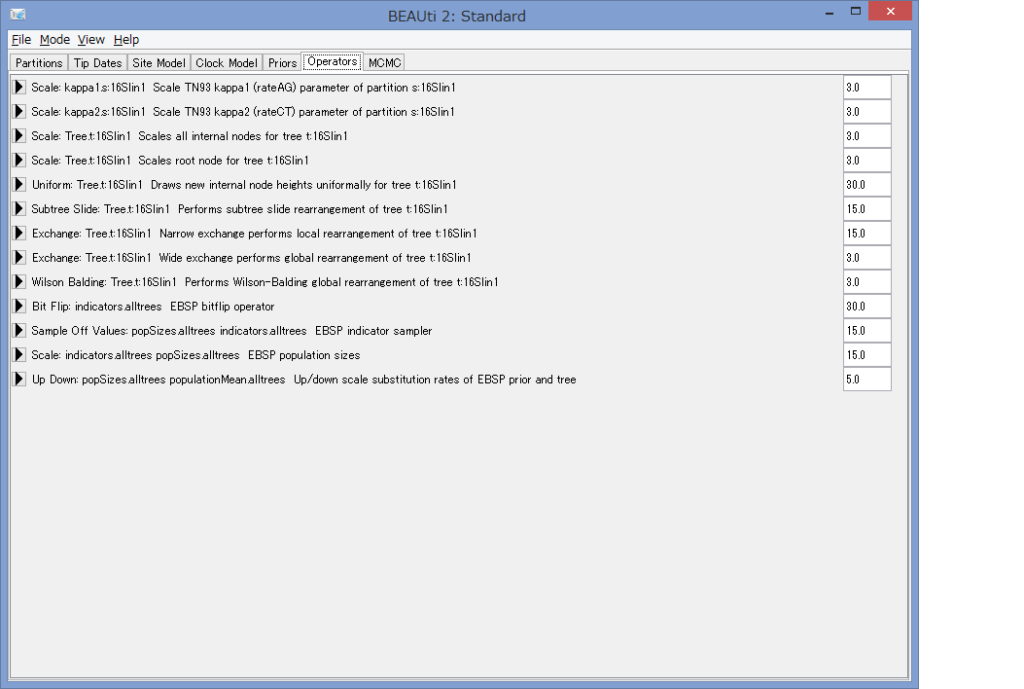
- もし複数の遺伝子データを使う場合は,Operatorsタブで,Bit Flip…, Sample off…, Scale…の値をそれぞれの数×30, 15, 15にするとよいようです.今回はデフォルトですが,もし二つの遺伝子を使う場合は60, 30, 30にするとよいでしょう.
- これで準備OKです.File→Saveでxmlファイルを保存しましょう(Sky.xmlとしましょう).
| MCMCによる解析(BEAST) |
BEASTでの作業
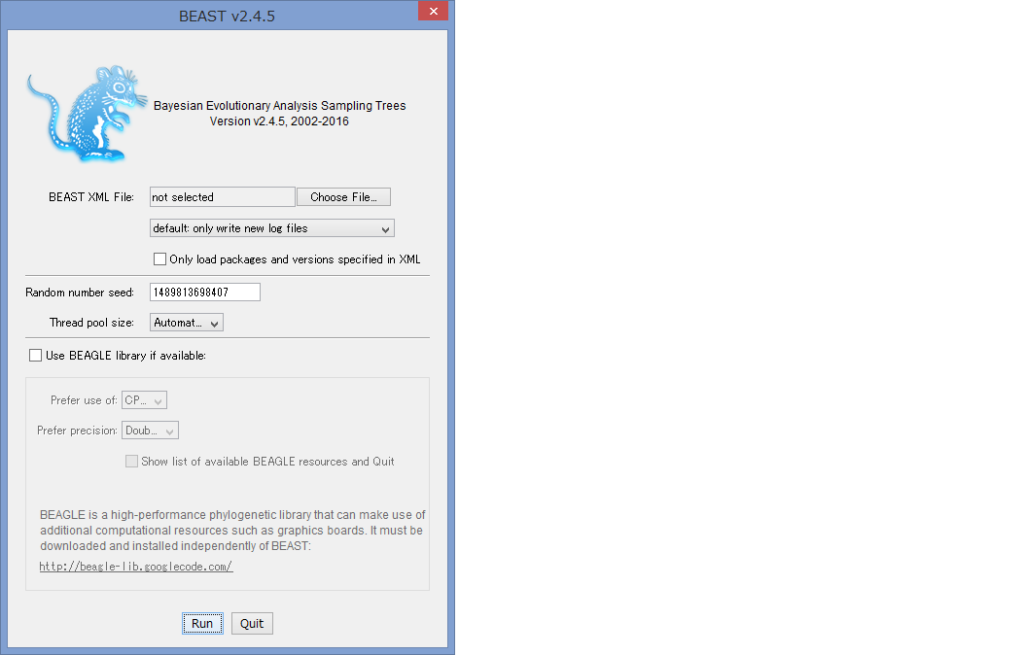
- BEASTを立ち上げ,Choose FileでSky.xmlを選ぶ.後はデフォルトでOKです.Runをクリックして解析を始めましょう.
- 上手くいけば,カリカリ解析が始まるはずです.苦労したファイルが走るのを見るとなんか感動しますよね.
- 解析が終了して,SkyフォルダにSky.logとEBSP.logが生成されればOKです.
| 集団推移の可視化(Tracer, R) |
Tracerでの作業
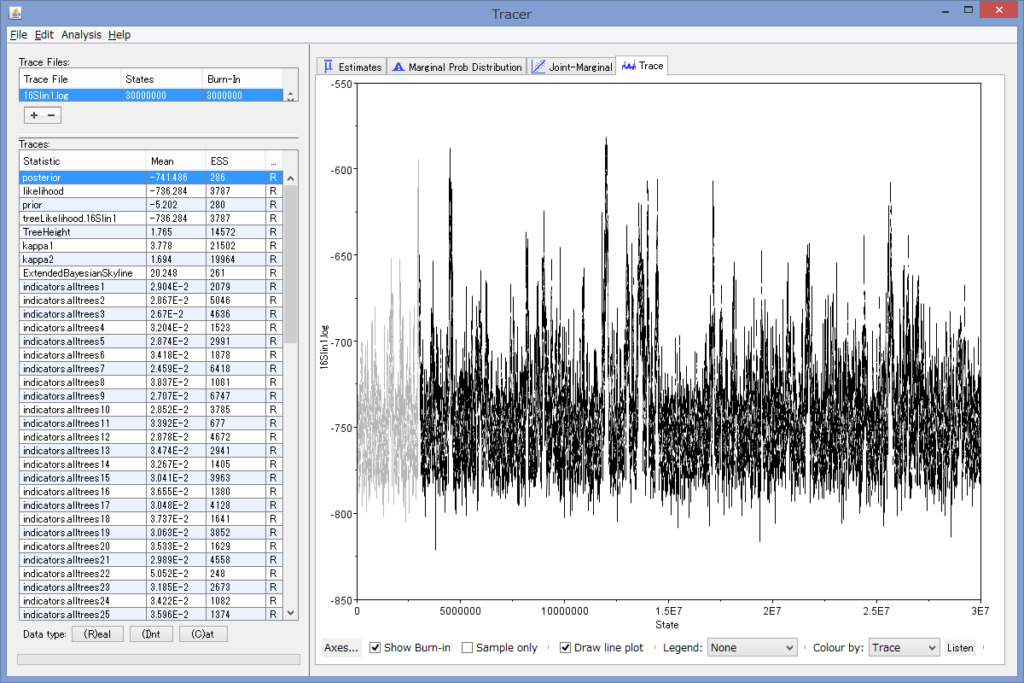
- Tracerを立ち上げ,Import Trace FileでSky.logを選ぶと,ファイルが読み込まれて,Traces:のところにlogの解析データが出力されます.
- このうち,ESSの値が重要なようで,これが200以下の項目がある場合はMCMCの連鎖回数を増やすとよいでしょう.
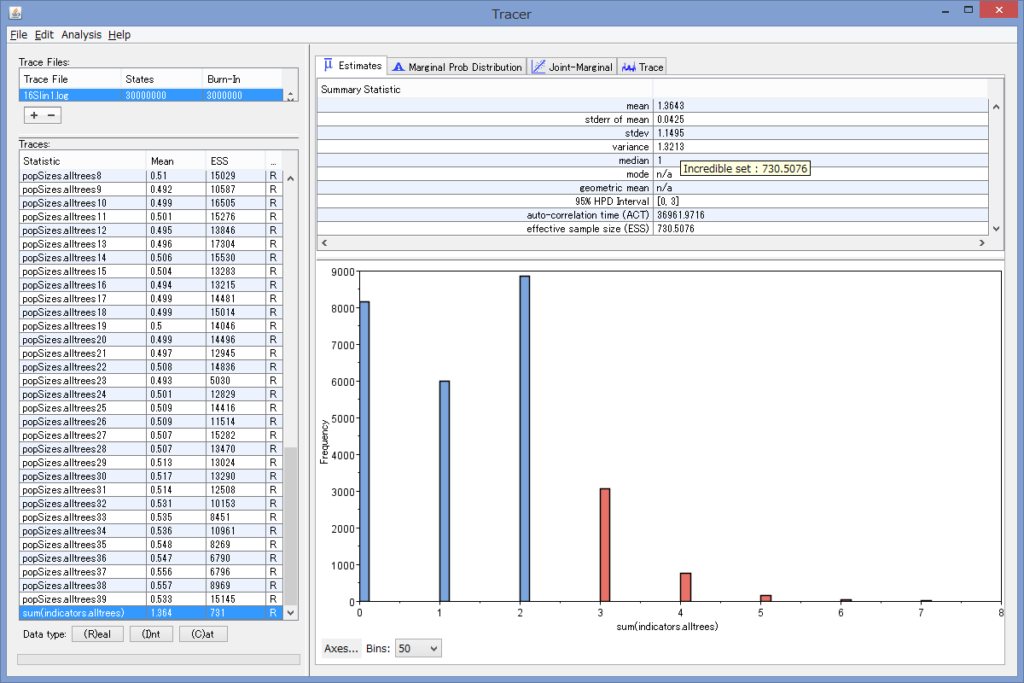
- 一番下のsum(indicators.alltrees)をクリックし,右側のEstimatesのタブをクリックすると,ヒストグラムが表示されます.
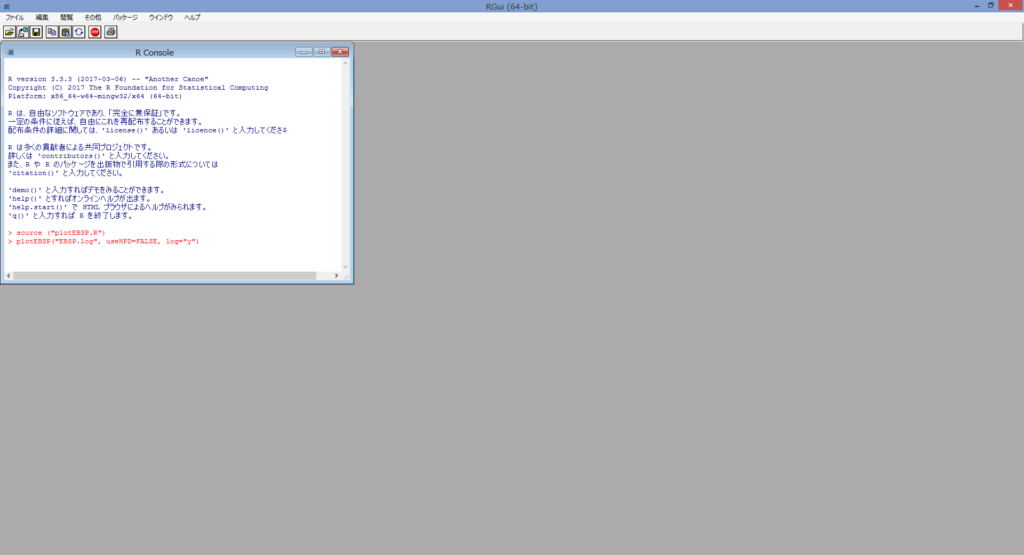
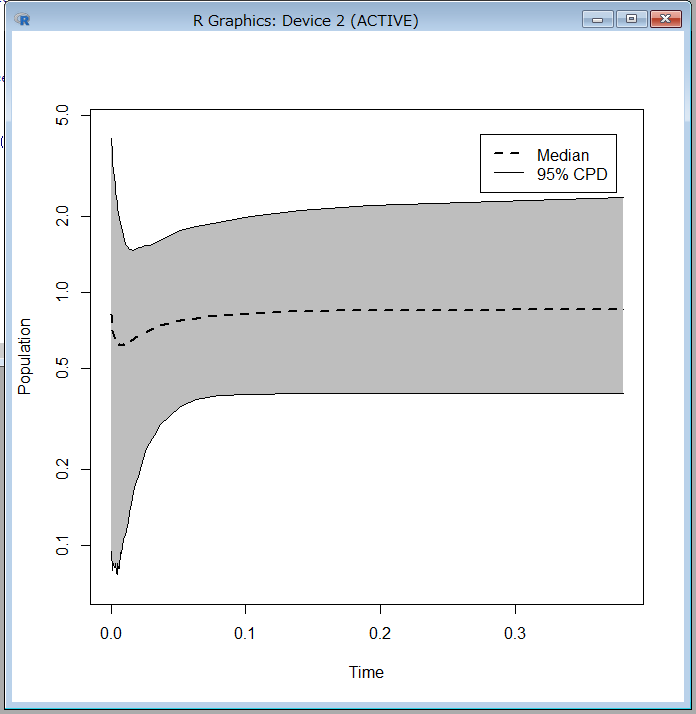
- Rを立ち上げ,Skyフォルダ内に移動し,以下のように入力すれば,少し計算時間があった後,集団の遷移図がプロットされます.
> source (“plotEBSP.R”)
> plotEBSP(“EBSP.log”, useHPD=FALSE, log=”y”)
- でました.横軸が年代(百万年)で,縦軸が集団サイズです.左が現在ですので,この集団はごく最近に少しだけ集団サイズの減少を経験しているようです.
- x軸の表示範囲を調整するには,Rに以下のように(0~0.03の範囲で表示する場合)入力します.
> plotEBSP(“EBSP.log”, useHPD=FALSE, xlim=c(0, 0.03))
今日はここまで.