MEGAでは塩基(アミノ酸)置換の進化モデルテストを行うことができます.
進化モデルとは,解析するデータセットの中における,
①塩基(アミノ酸)の存在頻度
②塩基(アミノ酸)同士の置換確率
の組み合わせです.
最尤法では,節間の置換を計算するので,この進化モデルの選択は非常に重要です.
まずはアライメントした配列を用意します.今回はCOI領域の配列です.
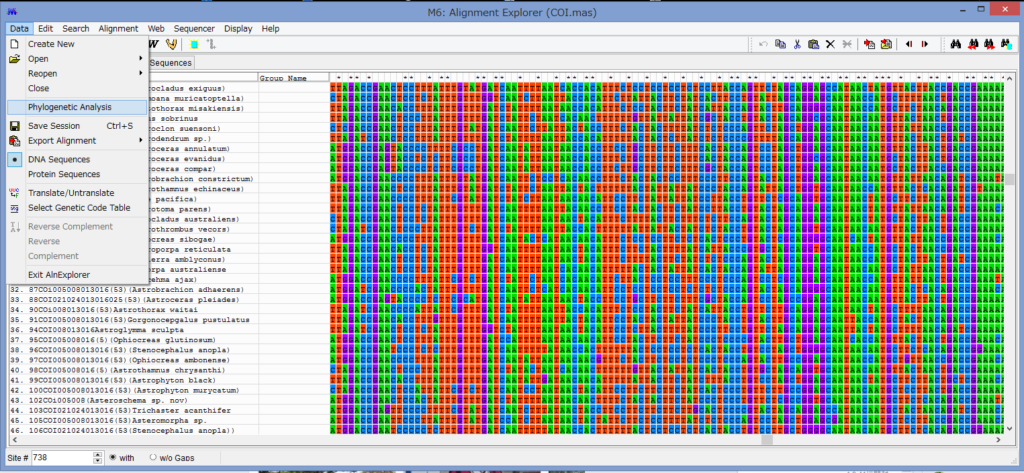
Data→Phylogenetic Analysisを選択します.
タンパク質のコーディング領域かどうかを聞かれます.
今回はCOIなのでYesです.
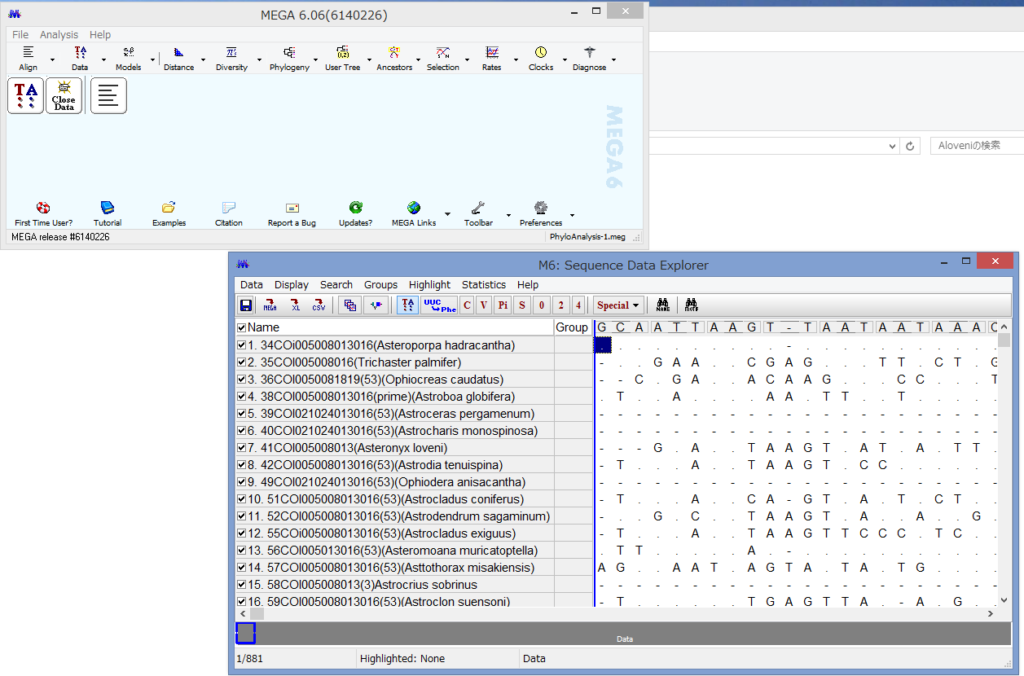
このようなウィンドウが現れたらOKです.アライメントの中から,塩基置換が起きている部分だけを取り出したものです.
このPhylogenetic analysisモードは,MEGAで系統解析を行うための基本モードです.
Models→Find Best DNa/Protain Models (ML)を選びます.
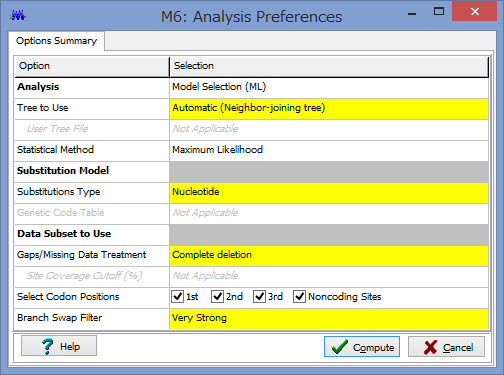
この画面でモデルテストのセッティングができます.
Tree of use :テストの際に使う系統樹を指定できます.通常,デフォルトのNJ法でOKです.
Substitution type:Nucleotide(塩基)やAmino acid(アミノ酸)が選べます.
Gaps/Missing Data Treatment:ギャップやミッシングデータの取り扱いが選べます.Complete deletionで,ギャップが一つでもある列は解析殻除きます.Partial Deletionで,比較する配列ごとにギャップの扱いを決めます.Use all siteでギャップもミッシングデータも全て扱います.
Select Codon Positions:チェックを外したコドンを解析から除きます.コーディング領域の場合,特に3rdコドンが他とモデルが異なる場合があったりするので,きちんと全てのコドンについてのモデルテストを行っておくことをお薦めします.
Branch Swap Filter:系統樹の枝長に対する厳密性を決めるオプションのようです.複雑なモデルを使うと,色んな塩基置換パターンを考慮に入れられる反面,その分の解析上の煩雑さも増えてしまいます.この煩雑さを枝長と考え,各モデルごとに枝長と尤度を比較し,尤度の上昇と枝長の少なさのバランスが最も良いものをベストのモデルとして選ぶようです.この枝長は系統樹の探索によって決められていくのですが,その際に系統樹の一部の枝を入れ替えます.この時の入れ替えの「大胆さ」を決めるのがこのオプションのようです.従って,Strongにすると枝長の入れ替えがより消極的になり,解析時間は短くなりますが,考慮する系統樹は少なくなります.よりWeakにすると,枝長の入れ替えが大胆になり,解析時間は長くなりますが,考慮する系統樹が多くなるようです.ですので,時間に余裕があるときはよりWeakにするとよいでしょう.
※個人的な見解ですので,間違っていたら教えてください.
さて,長くなりましたが,セッティングを終えてComputeを選択するとモデルテストが始まります.
モデルを一つ一つ試しています.終わるまで気長に待ちましょう.
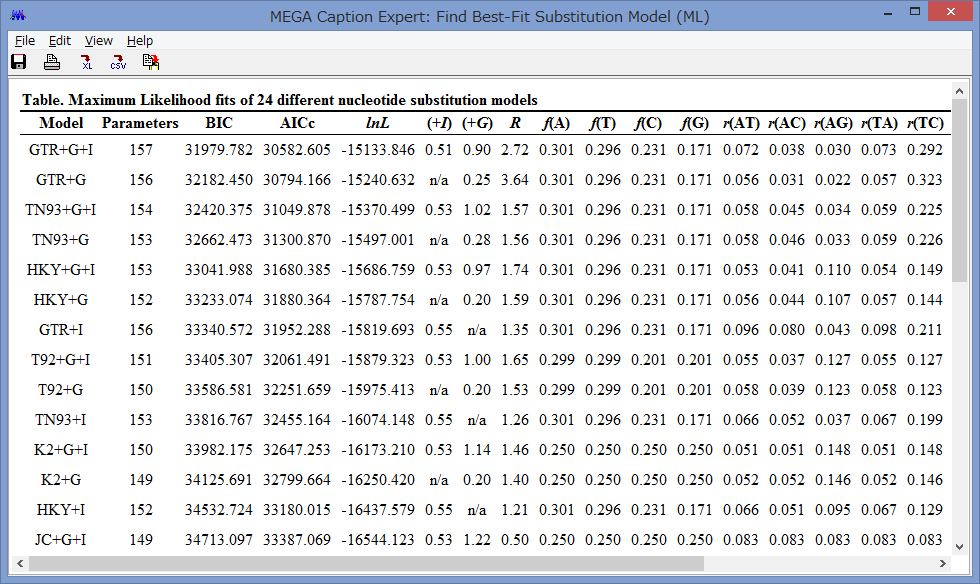
結果が出ました.注目すべきは,BIC, AICcです.これらの値が最も少ない(=尤度と煩雑さのバランスが最も良い)ものが,ベストなモデルとなります.
今回の解析では,GTR+G+Iがベストなモデルとして選択されました.他にも,f(A),f(T)…などはそのモデルの下での塩基の存在頻度で,r(AT)…などが置換確率となります.
このウィンドウの下の方に,それぞれのモデルに対する説明があります.
ここを参考にして,今後の最尤法解析などの際にパラメーターを設定します.
今日はここまで.